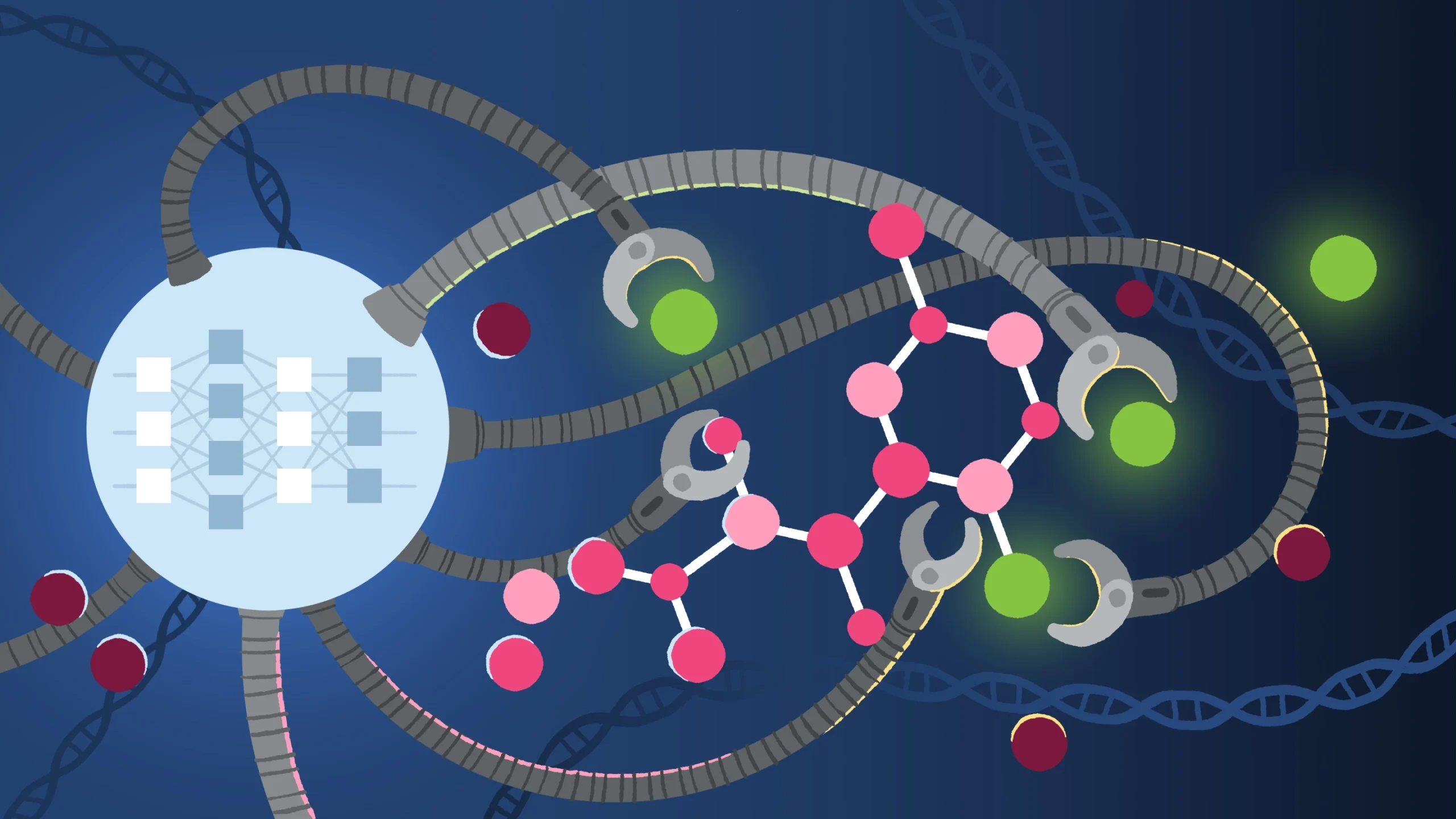
پیشرفت AlphaFold3، گامی فراتر از دستاوردهای قبلی در زمینه پیشبینی ساختار پروتئینها بهحساب میآید. نسخههای پیشین این الگوریتمها، اگرچه در پیشبینی ساختار پروتئینها موفق بودند، اما قادر به درک نحوه تعامل پروتئینها با سایر مولکولهای حیاتی نبودند. این موضوع بهدلیل آن بود که پروتئینها بهندرت بهتنهایی عمل میکنند و عملکرد آنها در تعامل با سایر مولکولها شکل میگیرد. در مقایسه مستقیم، به نظر میرسد AlphaFold3 از نظر دقت از RoseTTAFold All-Atom پیشی میگیرد.
پیشبینی دقیق نحوه چینش اجزای سازنده سلول
ابزارهای نوظهور هوشمصنوعی، گامی بلند
در جهت درک عمیقتر نحوه عملکرد سلولها برداشتهاند. الگوریتمهای پیشرفته یادگیری
عمیق مانند AlphaFold3
از شرکت DeepMind
گوگل، اکنون قادر به پیشبینی دقیق شکل و نحوه چینش مولکولهای حیاتی
مانند پروتئینها، DNA
و RNA
هستند. این پیشرفت، دریچهای نو بهسوی درک پیچیدگیهای ساختاری و عملکرد
سلولها میگشاید.
پروتئینها، بهعنوان ماشینهای مولکولی
سلول، نقشی اساسی در حفظ سلامت و عملکرد ارگانیسمها ایفا میکنند. شناخت دقیق ساختار
سهبعدی پروتئینها، کلید فهم نحوه عملکرد آنها در فرآیندهای مختلف سلولی و همچنین
درک مکانیسم بیماریها ناشی از اختلال در عملکرد پروتئینها است.
با توسعه الگوریتمهای جدید یادگیری ماشینی،
محققان گامی بلند در جهت پیشبینی دقیق ساختار سهبعدی پروتئینها و سایر مولکولهای
حیاتی برداشتهاند. این الگوریتمها قادرند شکل پیچیده مولکولها را با دقتی بیسابقه
پیشبینی کنند، گامی که پیش از این غیرقابلدسترسی به نظر میرسید.
در مقالهای که بهتازگی در مجله Nature منتشر شدهاست، شرکت DeepMind و Isomorphic Labs،
نسخه جدیدی از برنامه AlphaFold
بهنام AlphaFold3 را معرفی کردهاند. این الگوریتم
پیشرفته، نهتنها قادر به پیشبینی ساختار پروتئینها بهتنهایی است، بلکه میتواند
ساختار DNA،
RNA،
لیگاندها و سایر مولکولهای حیاتی را نیز بهطور دقیق پیشبینی کند. این قابلیت، به
محققان امکان میدهد تا نحوه تعامل این مولکولها با یکدیگر و نقش آنها در فرآیندهای
سلولی را بهطور دقیقتری درک کنند.
پیشرفت AlphaFold3، گامی فراتر از دستاوردهای قبلی در زمینه پیشبینی
ساختار پروتئینها بهحساب میآید. نسخههای پیشین این الگوریتمها، اگرچه در پیشبینی
ساختار پروتئینها موفق بودند، اما قادر به درک نحوه تعامل پروتئینها با سایر مولکولهای
حیاتی نبودند. این موضوع بهدلیل آن بود که پروتئینها بهندرت بهتنهایی عمل میکنند
و عملکرد آنها در تعامل با سایر مولکولها شکل میگیرد.
 John Jumper, who joined Google DeepMind in 2017, led the team that produced AlphaFold3, which can predict the structures of biomolecular complexes. “I can’t wait to see what [researchers] do with AlphaFold3,” he said
John Jumper, who joined Google DeepMind in 2017, led the team that produced AlphaFold3, which can predict the structures of biomolecular complexes. “I can’t wait to see what [researchers] do with AlphaFold3,” he said
پیشرفت AlphaFold3، گامی فراتر از دستاوردهای قبلی در زمینه پیشبینی ساختار پروتئینها
بهحساب میآید. نسخههای پیشین این الگوریتمها، اگرچه در پیشبینی ساختار پروتئینها
موفق بودند، اما قادر به درک نحوه تعامل پروتئینها با سایر مولکولهای حیاتی نبودند.
این موضوع بهدلیل آن بود که پروتئینها بهندرت بهتنهایی عمل میکنند و عملکرد آنها
در تعامل با سایر مولکولها شکل میگیرد.
جان جامپر، رهبر تیم AlphaFold در DeepMind در این باره میگوید: "هر بار که در مورد AlphaFold2 صحبت میکردم، میتوانستم حدس بزنم که چه سوالی مطرح
خواهد شد. افراد میپرسیدند که AlphaFold2 چگونه میتواند نحوه تعامل پروتئینها با DNA را پیشبینی کند؟ " جامپر اذعان میکرد که AlphaFold2 پاسخ روشنی برای این سوال نداشت.
اما AlphaFold3 با قابلیت پیشبینی ساختار و نحوه تعامل مولکولهای
مختلف، دریچهای نو بهسوی درک عمیقتر فرآیندهای سلولی و کشف راهحلهای نوین برای
درمان بیماریها میگشاید. این الگوریتم، ابزاری قدرتمند در دستان محققان برای رمزگشایی
از اسرار حیات و گامی بلند در جهت ارتقای سلامت بشر است.
AlphaFold3: فراتر از پروتئینها، بهسوی درک عمیقتر حیات
اما AlphaFold3 میتواند گامی فراتر بردارد. این الگوریتم پیشرفته،
بههمراه دیگر الگوریتمهای نوظهور یادگیری عمیق، فراتر از پیشبینی ساختار پروتئینها
رفته و چشماندازی چالشبرانگیزتر و مرتبطتر از زیستشناسی سلولی ارائه میدهد. این
الگوریتمها قادر به پیشبینی ساختار و نحوه تعامل طیف گستردهای از مولکولهای حیاتی
در سلولها هستند.
برندا روبنشتاین، دانشیار شیمی
و فیزیک در دانشگاه براون که در این مطالعات دخیل نبوده است، میگوید: "با AlphaFold3 میتوانیم به درک عمیقتر تعاملات پیچیدهای که در زیستشناسی
اهمیت دارند، دست پیدا کنیم. گویی قطعات پازل در حال کنار هم قرار گرفتن هستند و تصویر
بزرگتر از عملکرد سلولها آشکار میشود. "
پل آدامز، بیوفیزیکدان مولکولی
در آزمایشگاه ملی لارنس برکلی نیز که در این مطالعات شرکت نداشتهاست، معتقد است:
"درک این تعاملات مولکولی، بنیادی برای درک عملکرد بیولوژیکی است. هر دو الگوریتم
AlphaFold3 و RoseTTAFold پیشرفت چشمگیری در این زمینه داشتهاند. "
با وجود اینکه هر دو الگوریتم
محدودیتهایی دارند، و البته پتانسیل تکامل به ابزارهای پیشبینی قدرتمندتر را نیز
دارا هستند. در ماههای آینده، دانشمندان با آزمایش این الگوریتمها، کاربردها و مزایای
شگفتانگیز آنها را در زمینههای مختلف علمی و درمانی آشکار خواهند کرد.
هوشمصنوعی، انقلابی در زیستشناسی: رمزگشایی از ساختار پروتئینها
یادگیری عمیق، شاخهای از هوشمصنوعی
که از مغز انسان الهامگرفته شده، تحولی شگرف در عرصه زیستشناسی ایجاد کرده است. الگوریتمهای
پیچیدهای که با شبکههای عظیمی از گرههای اطلاعاتی (نورونها) ساختهشدهاند، گویی
مغزی مصنوعی را به وجود میآورند که قادر به یادگیری و حل مسائلی است که تا پیش از
این غیرقابل حل به نظر میرسیدند.
در این میان، پیشرفتهای چشمگیری
در زمینه پیشبینی ساختار پروتئینها بهدست آمده است. پروتئینها، مولکولهای پیچیدهای
هستند که نقش کلیدی در عملکرد سلولها و موجودات زنده ایفا میکنند. درک ساختار سهبعدی
پروتئینها، برای فهم چگونگی عملکرد آنها و در نهایت، توسعه داروها و درمانهای جدید
حیاتی است.
AlphaFold2، نقطه عطفی در این مسیر بود. این الگوریتم یادگیری
عمیق که در سال 2021 معرفی شد، دریچهای نو بهسوی دنیای ناشناخته ساختارهای پروتئینی
گشود و به ابزاری قدرتمند در دستان محققان تبدیل شد. از AlphaFold2 میتوان برای درک ساختار سلولها، مطالعه بیماریهایی
مانند سل و بسیاری موارد دیگر استفادهکرد.
علاوه بر AlphaFold2، الگوریتمهای پیشرفته دیگری مانندRoseTTAFold نیز توسط محققان توسعهیافتهاند. این الگوریتمها که
با الهام از AlphaFold2 ساختهشدهاند، قادر به پیشبینی
ساختار پروتئینها از روی توالی اسیدهای آمینهای آنها هستند.
این پیشرفتها، نویدبخش تحولات
عظیمی در زمینه زیستشناسی و پزشکی هستند. با درک عمیقتر از ساختار و عملکرد پروتئینها،
میتوانیم به راهحلهای نوینی برای درمان بیماریها، طراحی داروهای جدید و ارتقای
سلامت بشر دست پیدا کنیم.
از زمان معرفی AlphaFold2 و RoseTTAFold،
این دو الگوریتم پیشرفته یادگیری عمیق با قابلیتهای جدیدی بهروز شدهاند. RoseTTAFold Diffusion امکان طراحی پروتئینهای جدیدی
را فراهم میکند که در طبیعت یافت نمیشوند. AlphaFold Multimer نیز قادر به بررسی نحوه تعامل چندین پروتئین با یکدیگر
است.
اما چالشی بزرگ باقی ماندهبود:
درک نحوه تعامل پروتئینها با سایر اجزای سلول. به گفته جان جامپر، رهبر تیم AlphaFold در DeepMind،
"ما تاکنون موفق به پیشبینی ساختار پروتئینها بهتنهایی شدهبودیم، اما در مورد
نحوه ارتباط آنها با DNA، RNA و سایر مولکولهای حیاتی در سلول، اطلاعات کافی نداشتیم. "
موفقیت اولیه الگوریتمهای یادگیری عمیق در پیشبینی ساختار پروتئینها، مرهون دسترسی به حجم عظیمی از دادههای آموزشی بود. حدود 140 هزار ساختار پروتئینی معتبر در طول 50 سال در "بانک داده پروتئین" (Protein Data Bank) ذخیره شدهبودند. علاوه بر این، زیستشناسان بهطور فزایندهای در حال جمعآوری اطلاعات مربوط به ساختار مولکولهای کوچک، DNA، RNA و نحوه تعامل آنها با یکدیگر بودند.
جان جامپر در این باره میگوید:
"بزرگترین چالش در توسعه الگوریتم AlphaFold برای درک تعاملات پروتئینها با سایر مولکولهای سلولی، این بود که آیا دادههای
کافی برای آموزش دقیق الگوریتم در این زمینه وجود خواهد داشت یا خیر. "
خوشبختانه، دادههای کافی در دسترس
بود. در اواخر سال 2023، بی کر و جامپر نسخههای اولیه ابزارهای هوشمصنوعی جدید خود
را منتشر کردند و از آن زمان تاکنون بهطور مداوم در حال ارتقا و بهبود الگوریتمهای
خود بودهاند.
محمد القریشی، زیستشناس سیستمی
در دانشگاه کلمبیا که در هیچیک از این پروژهها دخیل نبوده است، میگوید: "هر
دو سیستم هوشمصنوعی بهدنبال پاسخ به یک سؤال واحد هستند، اما روشهای یادگیری عمیق
آنها بر پایه معماریهای متفاوتی بنا شدهاند. "
القریشی توضیح میدهد که تیم جامپر
از روشی بهنام "انتشار" (diffusion)
استفاده میکند. این فناوری که در بسیاری از سیستمهای هوشمصنوعی مولد غیر متنی مانند
Midjourney و DALL·E نیز به کار برده میشود، قادر به تولید آثار هنری بر
اساس دستورات متنی است. در این روش، بهجای پیشبینی مستقیم ساختار مولکولی و سپس اصلاح
آن، ابتدا تصویری تار از ساختار مورد نظر ایجاد میشود و سپس بهطور مکرر پالایش و
اصلاح میشود تا به نتیجه نهایی مطلوب دست پیدا کند.
RoseTTAFold All-Atom: پیشگویی ساختار زیستمولکولها و طراحی مولکولهای جدید
محمد القریشی، زیستشناس سیستمی
در دانشگاه کلمبیا، معتقد است که از نظر فنی، جهش بزرگی از RoseTTAFold به RoseTTAFold All-Atom صورت نگرفتهاست. بی کر، توسعهدهنده این الگوریتم، معماری اصلی RoseTTAFold را بهطور اساسی تغییر نداده، اما آن را با قوانین شناختهشده
تعاملات بیوشیمیایی بهروزرسانی کرده است.
برخلاف AlphaFold Diffusion، که از روش انتشار برای پیشبینی ساختارهای زیستمولکولی استفاده میکند،
RoseTTAFold All-Atom از این روش بهره نمیبرد. با این
حال، هوشمصنوعی بی کر از انتشار در فرآیند طراحی پروتئینها استفاده میکند. آخرین
نسخه این برنامه، RoseTTAFold
Diffusion All-Atom،
قادر به طراحی زیستمولکولهای جدیدی فراتر از پروتئینها نیز هست.
القریشی میگوید: "مزایای
استفاده از هوشمصنوعی مولد در زمینه زیستمولکولها فقط به طراحی پروتئین محدود نمیشود.
اگر بتوانیم از این روش برای طراحی مولکولهای کوچک نیز استفاده کنیم، تحولات شگفتانگیزی
در انتظار خواهد بود. "
رقابت AlphaFold3 و RoseTTAFold All-Atom:
کدامیک دقیقتر است؟
در مقایسه مستقیم، به نظر میرسد
AlphaFold3 از نظر دقت از RoseTTAFold All-Atom پیشی میگیرد. بهعنوان مثال، در تحلیلی که تیم گوگل
در مجله Nature منتشر کرده است، مشخص شده که AlphaFold3 در پیشبینی ساختار پروتئینهایی که با مولکولهای کوچکی
بهنام لیگاند (ligand) برهمکنش میکنند، حدود 76 درصد دقت دارد. این
در حالی است که دقت RoseTTAFold
All-Atom در این زمینه حدود 42 درصد و بهترین ابزارهای جایگزین موجود، 52 درصد است.
 The biochemist David Baker has spearheaded the development of RoseTTAFold, a leading protein-prediction AI system. He released an update that models biomolecular complexes just a couple of months before Google DeepMind released theirs
The biochemist David Baker has spearheaded the development of RoseTTAFold, a leading protein-prediction AI system. He released an update that models biomolecular complexes just a couple of months before Google DeepMind released theirs
دیوید بی کر، توسعهدهنده RoseTTAFold All-Atom، عملکرد پیشبینی ساختار AlphaFold3 را "بسیار چشمگیر و بهتر از RoseTTAFold All-Atom" میداند.
با این حال، محمد القریشی، زیستشناس
سیستمی در دانشگاه کلمبیا، معتقد است که ارقام ارائه شده بر اساس مجموعه دادههای محدودی
است که چندان چالشبرانگیز نیست. او انتظار ندارد که تمام پیشبینیهای مربوط به کمپلکسهای
پروتئینی امتیاز بالایی داشتهباشند و مطمئناً ابزارهای هوشمصنوعی جدید هنوز بهاندازه
کافی قدرتمند نیستند تا بهتنهایی از یک برنامه قوی کشف دارو پشتیبانی کنند، زیرا این
امر مستلزم درک عمیق محققان از برهمکنشهای پیچیده زیستمولکولی است.
القریشی میگوید: "با این
حال، این پیشرفتها قطعاً امیدوارکننده هستند و بهطور قابلتوجهی از روشهای قبلی
پیشرفتهتر هستند. "
پل آدامز، بیوفیزیکدان مولکولی
در آزمایشگاه ملی لارنس برکلی نیز با این نظر موافق است. او میگوید: "اگر کسی
ادعا کند که میتوان از این ابزارها برای توسعه دقیق داروها در آینده نزدیک استفادهکرد،
من با او موافق نیستم. هر دو روش در دقت خود محدودیتهایی دارند، [اما] این پیشرفتها
گامی بزرگ در مقایسه با آنچه قبلاً امکانپذیر بود، محسوب میشوند. "
AlphaFold3 و RoseTTAFold All-Atom: ابزارهای نوین کشف علمی
این دو مدل هوشمصنوعی، بهویژه
برای ایجاد پیشبینیهای اولیه که میتوان آنها را بهصورت تجربی یا محاسباتی آزمایش
کرد، بسیار مفید خواهند بود. فرانک اولمان، زیستشناس، پس از ملاقات با یکی از کارمندان
گوگل در مؤسسه فرانسیس کریک در لندن، فرصتی برای پیشآزمایش AlphaFold3 پیدا کرد. او میگوید که تصمیم گرفته است بهدنبال برهمکنش
پروتئین-DNA باشد که "واقعاً برای ما
گیجکننده بوده است". AlphaFold3 پیشبینیای ارائه داد که اکنون
آنها در حال آزمایش تجربی آن در آزمایشگاه هستند. اولمان میگوید: "ما قبلاً
ایدههای جدیدی را بهدست آوردهایم که واقعاً ممکن است مؤثر باشند. این یک ابزار کشف
شگفتانگیز است. "
با این حال، هنوز جای پیشرفت بسیاری
وجود دارد. RoseTTAFold
All-Atom در پیشبینی ساختار کمپلکسهای پروتئینها و مولکولهای کوچک، گاهی اوقات مولکولها
را در جایگاه صحیح در پروتئین قرار میدهد، اما جهتگیری آنها را بهدرستی پیشبینی
نمیکند. AlphaFold3 نیز گاهی اوقات کایرالیتی (جهتگیری
هندسی "چپدست" یا "راستدست") یک مولکول را بهاشتباه پیشبینی
میکند. همچنین، این الگوریتمها گاهی اوقات توهم ایجاد میکنند یا ساختارهای نادرست
را خلق میکنند.
علاوه بر این، هر دو الگوریتم
فقط تصاویری ثابت از پروتئینها و کمپلکسهای آنها تولید میکنند. در حالی که در واقعیت،
پروتئینها در داخل سلول پویا هستند و بسته به محیط میتوانند تغییر کنند: حرکت میکنند،
میچرخند و شکلهای مختلفی به خود میگیرند. پل آدامز، بیوفیزیکدان مولکولی، میگوید
که پرداختن به این موضوع چالشبرانگیز خواهد بود، عمدتاً بهدلیل کمبود دادههای آموزشی.
او میگوید: "بسیار عالی خواهد بود که تلاشهای هماهنگی برای جمعآوری دادههای
تجربی طراحی شده برای آگاهسازی از این چالشها وجود داشتهباشد. "
AlphaFold3: گامی بلند در علم زیستشناسی، با محدودیتهایی جدید
یکی از تغییرات عمده در محصول
جدید گوگل، عدم انتشار متنباز آن است. در حالی که کد پایه AlphaFold2 در دسترس عموم قرار داشت و به زیستشناسان اجازه میداد
تا الگوریتم را در آزمایشگاههای خود بازتولید و با آن کار کنند، کد AlphaFold3 بهطور عمومی قابل دسترس نخواهد بود.
محمد القریشی، زیستشناس سیستمی،
میگوید: "به نظر میرسد که آنها روش را با جزئیات توصیف میکنند. اما حداقل در
حال حاضر، هیچکس نمیتواند آن را اجرا کند و از آن مانند AlphaFold2 استفاده کند. این یک قدم بزرگ به عقب است. البته ما
سعی خواهیم کرد آن را بازتولید کنیم. "
با این حال، گوگل برای افزایش
دسترسی به این ابزار، سرور AlphaFold جدیدی را به زیستشناسانی که
از AlphaFold3 استفاده میکنند، ارائه میدهد.
فرانک اولمان، زیستشناس، میگوید پیشبینی ساختارهای زیستمولکولی به قدرت محاسباتی
زیادی نیاز دارد. حتی در مؤسسهای مانند Francis Crick که از خوشههای محاسباتی با عملکرد بالا برخوردار است،
حدود یک هفته طول میکشد تا نتیجهای بهدست آید. از طرفی سرورهای قدرتمند گوگل میتوانند
در عرض پنج دقیقه پیشبینی کنند و دانشمندان سراسر جهان میتوانند از آنها استفادهکنند.
اولمان میگوید: "این قرار است تحقیقات پیشبینی پروتئین را کاملاً دموکراتیزه
کند. "
تأثیر واقعی این ابزارها تا ماهها
یا سالها، زمانی که زیستشناسان شروع به آزمایش و استفاده از آنها در تحقیقات خود
کنند، مشخص نخواهد شد. علاوه بر این، این ابزارها به تکامل خود ادامه خواهند داد. دیوید
بی کر، توسعهدهنده RoseTTAFold
All-Atom، میگوید
که گام بعدی برای یادگیری عمیق در زیستشناسی مولکولی، "بالا رفتن از نردبان پیچیدگی
بیولوژیکی" و فراتر رفتن از کمپلکسهای زیستمولکولی پیشبینیشده توسط AlphaFold3 و RoseTTAFold All-Atom است.
جان جامپر، رهبر تیم AlphaFold در DeepMind، میگوید: "چیزهای بسیار بیشتری برای فهمیدن وجود دارد. تازه شروع کار است. "